Large genome annotation
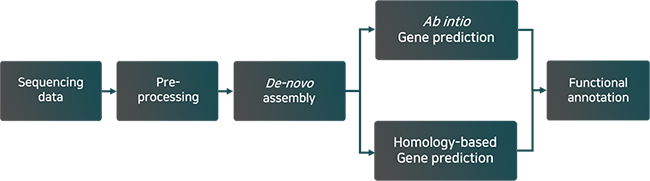
Plant genome annotation 분석은 식물 유전체에서 유전자의 구조적 정보를 예측하는 Gene modeling과 유전자의 기능을 규명하는 Functional annotation 분석을 말합니다. Gene modeling 방법은 크게 두 가지로 나눌 수 있는데 ab intio 방법과 homology-based 방법입니다. ab initio 방법은 Augustus, Fgenesh 등의 프로그램을 통해 수학적 알고리즘으로 유전자 모델을 트레이닝하고, 해당 결과로 유전자의 exon과 intron 영역 및 위치를 예측하는 방법입니다.
또한 Homology-based 방법은 BLAT과 같은 tool을 이용해 유전체에 어셈블리 서열을 alignment 하여 일정 기준 이상의 homology를 갖는 위치를 예측하고, align된 위치를 유전자의 유전체 내 위치로 예측하는 방법입니다. 이 두 방법의 결과를 잘 결합하여 사용한다면 정제된 유전체의 annotation 정보를 얻을 수 있습니다. 또한 잘 연구된 protein DB과의 서열 유사성을 이용해 Functional annotation 분석을 할 수 있습니다.
또한 Homology-based 방법은 BLAT과 같은 tool을 이용해 유전체에 어셈블리 서열을 alignment 하여 일정 기준 이상의 homology를 갖는 위치를 예측하고, align된 위치를 유전자의 유전체 내 위치로 예측하는 방법입니다. 이 두 방법의 결과를 잘 결합하여 사용한다면 정제된 유전체의 annotation 정보를 얻을 수 있습니다. 또한 잘 연구된 protein DB과의 서열 유사성을 이용해 Functional annotation 분석을 할 수 있습니다.
Work Flow
Result Examples
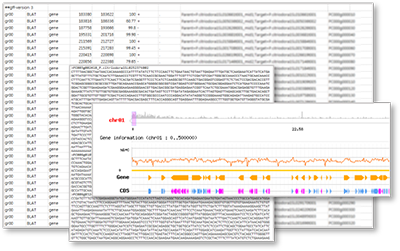 Sequence, Gene track 및 genome browser
Sequence, Gene track 및 genome browser
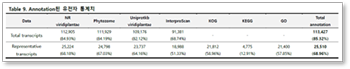 Functional annotation 결과 통계치
Functional annotation 결과 통계치
관련 분석사례
총 6건- 갯질경 genome assembly 및 annotation
- 애멸구 genome annotation
- 클로렐라 HS2 genome annotation
- 극지클로렐라 genome annotation
- 에뜰리아 genome annotation
- 나노클로롭시스 genome annotation


